고정 헤더 영역
상세 컨텐츠
본문
개발 동기
이전에 EigenRand라고 Eigen용 난수생성기를 개발한 적이 있는데요, 왜 다변량 분포(multivariate distribution) 지원은 없냐는 문의가 종종 들어오더라구요. 곰곰히 생각해보니 벡터와 행렬을 다루는 라이브러리에서 다변량 분포를 전혀 지원하지 않는게 이상해보여서 추가하기로 마음먹었습니다.
현재 C++표준에는 다변량 분포와 관련된 함수가 없기 때문에 통계/과학 연산을 위해서 널리 쓰이고 있는 Python 라이브러리인 scipy를 참고로하여 추가할 기능들을 정리해보았습니다. 대표적으로 통계학 분야에서 자주 쓰이는 다변량 분포에는 다음과 같은 것들이 있다고 합니다.
- 다변량 정규분포(Multivariate Normal Distribution)
- 다항 분포 (Multinomial Distribution)
- 디리클레 분포(Dirichlet Distribution)
- 위샤트 분포(Wishart Distribution)
- 역 위샤트 분포(Inverse Wishart Distribution)
위의 세 개는 N차원짜리 벡터를 생성하는 분포이고, 아래 두 개는 NxN 모양의 행렬을 생성하는 분포입니다. 이 분포들은 모두 Python SciPy에 구현되어 있으므로 이 Python 코드와 이 Fortran 코드를 베이스로 하여 C++ EigenRand용 구현체를 구현할 수 있었습니다.
성능 비교
레퍼런스로 삼은 Python scipy 모듈과의 속도 비교를 실시해봤습니다. Python과 C++의 비교라 불공정해보지만, 사실 SciPy 및 NumPy 내부 연산들은 모두 C 혹은 Fortran으로 작성되어 있으므로, 이를 wrapping하는데 발생한 약간의 오버헤드만 제외하면, 그렇게까지 불공정한 비교는 아닐 겁니다.
괄호 속의 숫자는 추출하는 표본의 차원수이며 multinomial에서는 속도에 중요한 영향을 미치는 시행횟수 t도 함께 표시했습니다. 각각 1만번씩 표본을 추출하는데 걸린 시간을 20번 측정해 평균을 구했으며, 단위는 ms입니다. 테스트 환경은 Intel(R) Xeon(R) Platinum 8171M (Ubuntu 16.04, gcc7.5)입니다.
| Python3.6 + scipy1.5 + numpy1.19 |
EigenRand (No Vect.) |
(SSE2) | (SSSE3) | (AVX) | (AVX2) | |
|---|---|---|---|---|---|---|
| Dirichlet(4) | 6.47 | 6.60 | 2.39 | 2.49 | 1.34 | 1.67 |
| Dirichlet(100) | 75.95 | 189.97 | 66.60 | 72.11 | 38.86 | 34.98 |
| InvWishart(4) | 140.18 | 7.62 | 4.21 | 4.54 | 3.58 | 3.39 |
| InvWishart(50) | 1510.47 | 1737.4 | 697.39 | 733.69 | 604.59 | 554.006 |
| Multinomial(4, t=20) | 3.32 | 4.12 | 0.95 | 1.06 | 1.00 | 1.03 |
| Multinomial(4, t=1000) | 3.51 | 192.51 | 35.99 | 39.58 | 27.84 | 35.45 |
| Multinomial(100, t=20) | 69.19 | 4.80 | 2.00 | 2.20 | 2.28 | 2.09 |
| Multinomial(100, t=1000) | 139.74 | 179.43 | 49.48 | 56.19 | 40.78 | 43.18 |
| MvNormal(4) | 2.32 | 0.96 | 0.36 | 0.37 | 0.25 | 0.30 |
| MvNormal(100) | 49.09 | 57.18 | 17.17 | 18.51 | 10.82 | 11.03 |
| Wishart(4) | 71.19 | 5.28 | 2.70 | 2.93 | 2.04 | 1.94 |
| Wishart(50) | 1185.26 | 1360.49 | 492.91 | 517.44 | 359.03 | 324.60 |
SciPy 대비 속도향상을 그래프로 그려보면 아래와 같습니다.
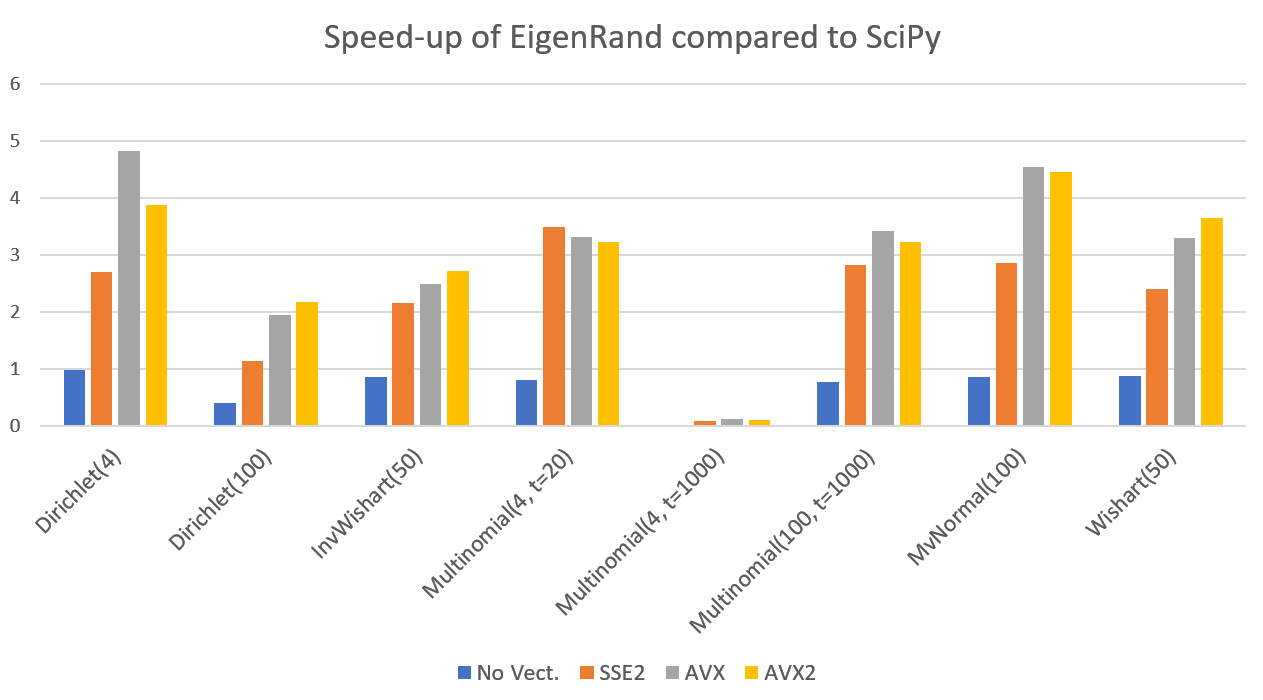
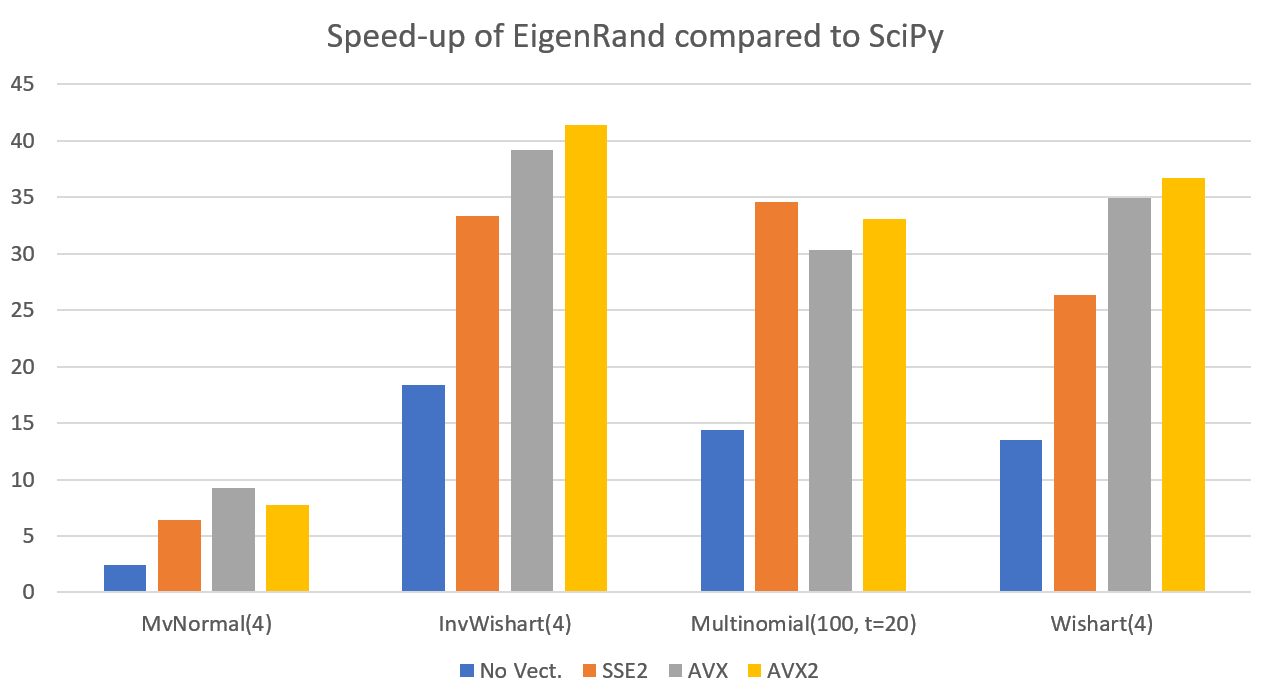
Vectorization을 사용하지 않았을때는 대부분 SciPy와 비슷한 속도가 나옵니다. 이를 보면 SciPy의 C 및 Fortran 구현이 vectorization을 활용하지 못하고 있다는 것을 간접적으로 확인할 수 있습니다. 반면 Vectorization을 활용하는 SSE2나 AVX, 혹은 더 최신의 AVX2의 결과를 보면 최소 2~3배, 빠르면 30배 가까이도 차이가 나는 걸 볼 수 있습니다. 당연히 Vectorization만으로 30배까지 차이가 나는것은 아니고, Python wrapper의 오버헤드 때문에 차이가 더 극적으로 발생한 것으로 보입니다.
예외적으로 낮은 차원의 벡터를 생성하는 Multinomial의 경우 Python보다 비효율적인 결과를 보여주었는데, 이는 SciPy의 multinomial함수가 binomial을 반복호출하는 것으로 결과를 생성하는 반면, EigenRand에서는 이 과정이 Vectorization이 불가하여 불가피하게 직접 표본을 시행횟수 t만큼 추출하는식으로 구현했기 때문에 발생한 문제입니다. 이 부분은 조금 더 개선이 필요해보입니다.
최신 버전의 EigenRand는 github에서 다운 받을 수 있습니다. 자세한 실험결과는 documentation에서 볼 수 있구요.
후기
흔히 C에 비해 Fortran이 수식계산에 빠르다고 널리 알려져 있고, 또 이 때문에 오래된 Fortran코드를 지금도 계속 쓰고 있는 경우가 많다고 합니다. 이번에 SciPy의 다변량 분포 구현을 C++로 옮겨보면서 느낀 점은 아마 vectorization을 고려하여 작성된 C++코드는 Fortran에 비해 느리지 않다는 것입니다 (더 빠를수도 있다고 봅니다). 아마 과학/수식 계산에 C가 느리다는 편견도 대량 수치 연산을 수행할때 Vectorization 최적화에 맞지 않게 코드를 작성해서 그런게 아닐까 싶더라구요. '바퀴를 재발명하지 말라'는 개발 쪽의 격언이 있긴 하지만, 종종 오래된 바퀴는 재발명해봄직하네요. 오래된 코드를 현재 아키텍처에 맞춰 다시 최적화할 수 있는 방법을 배울 수 있는 좋은 기회가 아닐까 싶습니다. 오래된 구현체를 C++로 옮겨보니 재미있네요.
'프로그래밍' 카테고리의 다른 글
| [C++] EigenRand: Eigen용 Random Library 개발 (0) | 2020.06.27 |
|---|---|
| 심심해서 해보는 딥러닝을 이용한 악기 소리 분류 (36) | 2019.12.02 |
| [Python] Segmented Least Squares를 이용해 구간 나누기 (0) | 2019.02.27 |
| [c++] 빠른 log sigmoid 계산 (0) | 2019.01.02 |
| [Python] 임의의 웹 페이지에서 텍스트를 추출하기 (1) | 2018.11.04 |
| [Python] 호환용 한자를 통합 한자로 변환하기 (2) | 2018.10.28 |


댓글 영역